检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
伴随着现代医学技术的发展,人类的平均寿命不断延长,老龄化逐渐成为世界各国所关注的重要问题,人们迫切希望深入了解衰老机制以及发展抗衰老的新方法。以往的衰老研究主要采用生物的实足年龄(Chronological Age)代表个体衰老程度。但是,即使在年龄相仿的一群人中,一些人外表更年轻也更有活力,这意味着个体的实足年龄并不能代表其真实衰老情况,即生物学年龄(Biological Age)。另外,实足年龄也无法用于评估抗衰老方法的干预效果。因此,如何量化个体衰老是衰老研究和评估衰老干预效果的关键性问题之一。
2013年,Steve Horvath以DNA甲基化为基础成功构建了第一个“衰老时钟”。在此之后,基于甲基化的衰老量化方法逐渐被广泛应用。然而,由于衰老过程的复杂性,单一类型的衰老时钟不一定能完整刻画个体衰老程度,组织和细胞的转录组变化也是衰老的重要标志之一,提示转录组数据可能被用于衡量衰老程度。使用单细胞转录组技术对衰老问题进行探索是近年来衰老研究领域的重要方向,因此,通过构建单细胞水平的衰老时钟模型,可以让我们更精细地衡量细胞衰老程度和细胞对于抗衰老方法响应的效果和异质性。
近日,js33333金沙线路检测李程课题组与卫生勤务与血液研究所陈河兵课题组合作,在Genome Research期刊在线发表了题为A transcriptome-based single-cell biological age model and resource for tissue-specific aging measures的研究论文。该研究利用基因表达调控在衰老过程中的变化,构建了一种基于单细胞转录组数据的生物学年龄估计模型(Single-cell aging-level estimator, SCALE)。

SCALE以衰老相关基因的表达量为基础,评估单细胞的衰老水平和细胞之间的衰老差异性。研究者首先通过文献挖掘,整理了以往研究已经报道的衰老相关基因并建立了Aging Map数据库。接着对小鼠衰老细胞图谱(Tabula Muris Senis)的单细胞转录组数据进行分析,进一步筛选出组织特异性的衰老基因。基于这些衰老基因集合,通过弹性网络回归方法构建了单细胞生物学年龄估计器(SCALE)。应用于不同单细胞数据集后,SCALE 估计的生物学年龄不仅与实足年龄显著相关,也与其他衰老相关指标(如体细胞突变数目、衰老相关疾病进展等)紧密相关,证明SCALE具有在单细胞层面区分衰老状态的能力。有趣的是,研究者发现同一组织器官内的不同细胞类型往往显示出不同的衰老程度,并且不同组织器官之间也存在不完全一样的细胞类型特异性衰老模式。
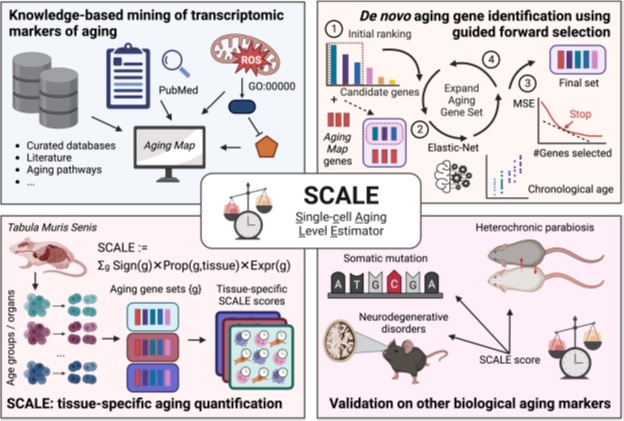
SCALE方法的分析流程示意图
另外,研究者发现SCALE可以用于评估抗衰老方法的干预效果。异体共生模型(heterochronic parabiosis)和热量限制(calorie restriction)是目前在模式生物中发现的两种有效干预衰老的手段。将SCALE应用于这些手段干预前后的转录组数据,均能精准预测干预组衰老状态的改变。
SCALE与现有的同类方法相比表现出良好的性能。由于SCALE充分利用了以往研究中的先验生物学知识,不仅提高了其可解释性,而且在面对单细胞组学数据中常见的批次效应、数据稀疏等问题时,SCALE能够保持高度的稳健性。此外,SCALE也表现出较好的可扩展性,当使用人类和大鼠中的同源基因替换其所用的小鼠衰老基因集时,SCALE可被用于估计相应物种单细胞水平的生物学年龄。
综上, SCALE方法不仅可以在单细胞水平预测生物学年龄、评估抗衰老方法的干预效果,而且能识别单细胞数据中的衰老细胞群体,为衰老机制和衰老细胞清除治疗的相关研究提供启发。
js33333金沙线路检测元培学院2018级本科生毛澍霖和js33333金沙线路检测2016级本科生苏嘉昱是该研究的共同第一作者。js33333金沙线路检测李程研究员和卫生勤务与血液研究所陈河兵副研究员是共同通讯作者。该研究得到了卫生勤务与血液研究所的伯晓晨研究员、js33333金沙线路检测中国卫生发展研究中心姚尧副教授的指导和帮助。
相关资源链接:
l Aging Map数据库(通过文献挖掘与整理的衰老相关基因集合):http://sysomics.com/AgingMap/
l SCALE软件包:https://github.com/ChengLiLab/SCALE